Leçon: Chaîne de traitement (workflow) en machine learning¶
Les différentes étapes¶
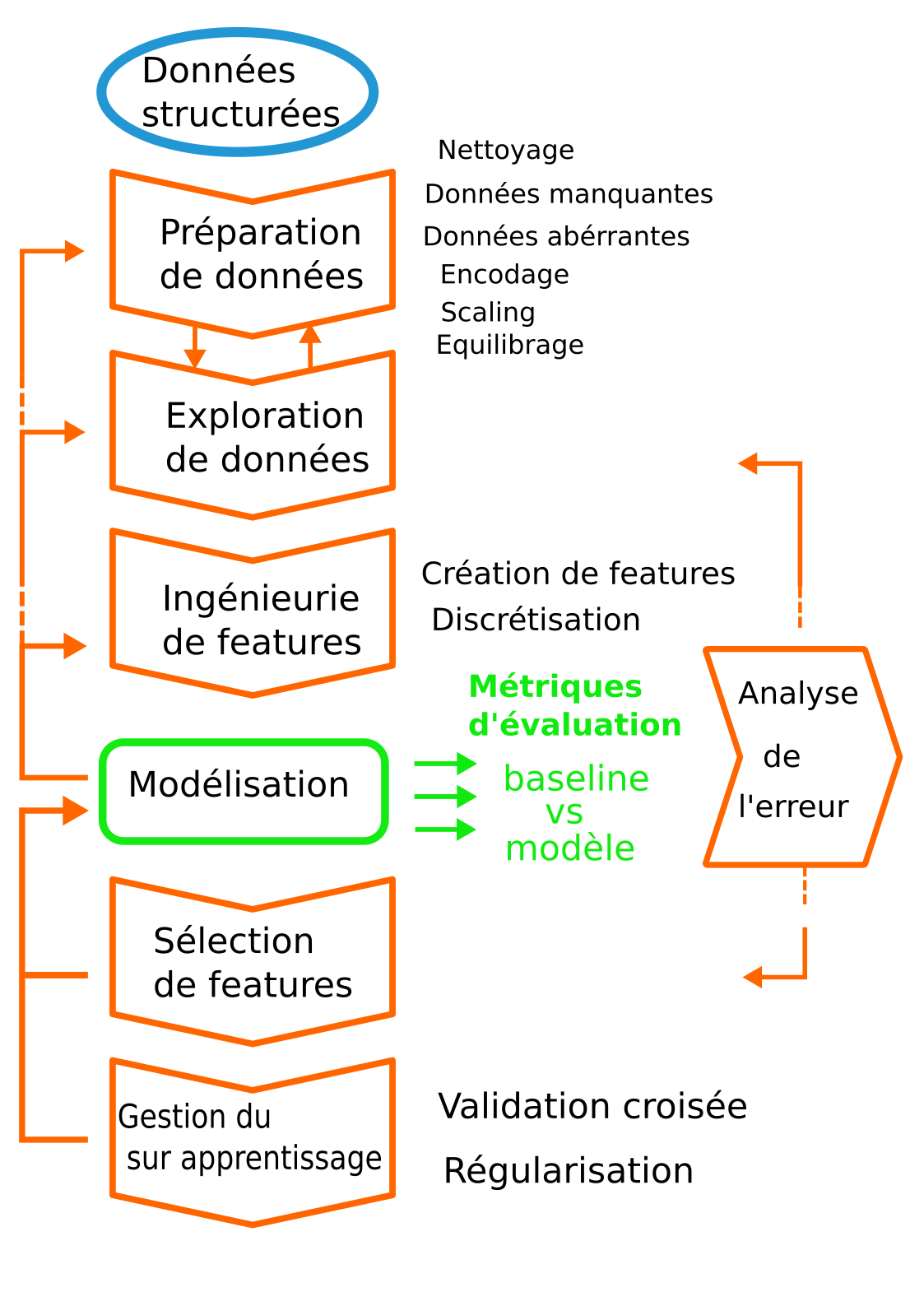
Bonnes pratiques (minimales) pour la construction de votre chaine de traitement¶
Organiser votre code de manière modulaire pour faciliter le lancement d'un grand nombre d'itérations
Sauvegarder les résultats de vos traitements : data set pré-traité (par ex avec pandas), modèle entrainé : préférez joblib pour vos modèles entrainés (ou utiliser la librairie généraliste pickle de pyhton)
joblib.save(model, path)
joblib.dump(model, path)
Enregistrer l'évolution des performances au fil des itérations: Par exemple avec mlflow
Etapes de traitement quasi-indispensables (pseudo-code)¶
Utiliser la méthode hold-out¶
X_train, X_test, y_train, y_test = train_test_split(X,y, test_size=..., random_state=...)
# fixer le random state à une valeur entière pour la reproductibilié
Pour apprendre et appliquer une transformation (souvent pendant le pré-traitement)¶
transformer = NomTransformation(parametres)
transformer.fit(X_train)
transformer.transform(X_train)
⚠️ Attention au risque de data leakage ☢️: Ne pas appliquer de transformation apprise en dehors du jeu d'apprentissage !
On apprend les transformation uniquement sur le jeu d'apprentissage, et on applique ces transformations à la fois sur celui d'apprentissage et de test
Exemple : appliquer un RobustScaler aux données¶
from sklearn.preprocessing import RobustScaler
X_train = [[ 1., -2., 2.],
[ -2., 1., 3.],
[ 4., 1., -2.]]
transformer = RobustScaler().fit(X_train)
transformer.transform(X_train)
array([[ 0. , -2. , 0. ],
[-1. , 0. , 0.4],
[ 1. , 0. , -1.6]])
Pour entrainer un modèle¶
model = NomDuModele(parametres,hypermarametres)
model.fit(X_train,y_train) # apprentissage supervisé
model.fit(X_train) # apprentissage non supervisé
Attention à toujours faire une optimisation ! ⚠️⚠️¶
Une erreur classique est d'entraîner, par facilité, un modèle avec les paramètres par défaut.
❌❌❌❌❌
model = NomDuModele()
model.fit()
❌❌❌❌❌
Ca n'est pas une bonne approche, car les paramètres utilisés par défaut ne donnent généralement pas des performances oet/u une intreprétabilité optimale, et peuvent même tendre au sur apprentissage (en particulier les méthodes basées sur les arbres de décision)
Vous devez TOUJOURS optimiser votre modèle pour trouver les paramètres les optimaux possible à utiliser lors de l'entrainement:
- ✅ soit en utilisant un optimiseur spécifique à votre modèle : Les
moindres carrés(pour la régression) , ladescente de gradient(réseaux de neurones et méthodes de boosting) - ✅ soit en utilisant des méthodes d'optimisation généralistes ! par exemple en utilsant des packages dédiés comme Optuna
- ✅ sinon, a minima avec des méthodes brute force comme le
GridSearchCV
Si vous n'avez pas de méthode d'optimisation spécifique: utilisez les méthode de type GridSearchCV¶
Si le modèle utilisé ne possède pas d'algorithme d'optimisation qui lui est applicable (par ex, la méthode des moindres carrés, applicables à la régression linéaire), on peut utiliser par défaut une méthode de type GridSearchCV
L'idée générale est de définir une grille de paramètre dans laquelle votre modèle sera entrainé avec des combinaisons de paramètres différentes, sur une partition cross validée de votre jeu de donnée d'apprentissage
On définit au préalable, une grille de paramètres à tester :
param_grid = {
"param_continu_1": [val1,val2,...]
"param_continu_2": [val1,val2,...]
"param_discret_1": ("val1","val2")
...
}
Puis on peut entraîne notre modèle, avec différentes stratégies pour le choix des combinaison des paramètres utilisés
Pour le GridSearchCV¶
On entraîne notre modèle de manière exhaustive pour chaque combinaison de paramètres de la grille
search = GridSearchCV(model, param_grid, refit=True, cv=5, ...)
search.fit(X_train, y_train)
Pour le HalvingSearchCV¶
Dans cette version, une sélection de paramètres (candidats) est entrainé de manière compétitive en plusieurs itérations. A chaque itération, seuls les meilleurs candidats sont sélectionnés et les ressources qui leur sont allouées pour chaque entraînement (en général des échantillons du dataset) sont multipliées par un facteur p.
Pour plus de détails voir le user guide
Il faudra fixer certains hyper-paramètres additionnels controllant le type de ressources, le nombre de candidats initial et le facteur utilisé :
search = HalvingGridSearchCV(model, param_grid, ressource='n_samples', n_candidates='exhaust', factor=3)
search.fit(X_train, y_train)
Pour le RandomizedSearchCV ou le HalvingRandomSearchCV¶
On entraîne notre modèle en tirant au hasard les paramètres continu de la grille
param_grid = {
"param_continu_1": scipy.stats.expon(loc=0, scale=100)# ici on tire au hasard des nombres entre 0 et 100 suivant une loi exponentielle
"param_discret_1": ("val1","val2")
...
}
search = RandomizedSearchCV(model, param_grid, refit=True, cv=5, random_state=42)
search.fit(X_train, y_train)
search = HalvingRandomSearchCV(model, param_grid, resource='n_samples', n_candidates='exhaust', factor=3)
search.fit(X_train, y_train)
Une fois l'optimisation terminée, on peut accéder à plusieurs attributs intéressants, en particulier les paramètres et le score de l'itération du modèle le plus performant:
search.best_params_
search.best_score_
ou encore au modèle ré-entrainé avec les meilleurs paramètres :
search.best_estimator_
Evaluer un modèle entraîné¶
une fois entrainé, il est possible de faire des prédictions et de calculer un score :
model.predict(X_test) # predictions sur le jeu de test
model.predict_proba(X_test) # uniquement pour certains modèles
model.score(X_test,y_test) # score calculé sur le jeu de test
la méthode .score calcule une métrique par défaut (accuracy pour la classification, $R^2$ pour la régression), mais il est possible de la changer, en appelant une métrique pré-definie dans scikit-learn:
par exemple si je souhaite utilise l'AUC :
from sklearn.metrics import auc
auc(X_test,y_test)
Représenter une courbe d'apprentissage¶
La courbe d'apprentissage est votre meilleur outil pour monitorer l'état de votre modèle dans le compromis biais/variance
train_sizes, train_scores, test_score = learning_curve(model, X, y)
plt.plot(train_sizes, np.mean(train_scores, axis=1), label="Training")
plt.plot(train_sizes, np.mean(test_scores, axis=1), label="Validation")
Vous pouvez utilisez la nouvelle classe LearningCurveDisplay pour tracer les learning curve plus facilement :
Il suffit de spécifier le modèle et des paramètres pour la représentation, le (.fit et le .predict) sont calculés automatiquement par scikit-learn :
params = {
"X": X_train,
"y": y_train,
"train_sizes": np.linspace(0.1, 1.0, 5),
"cv": ShuffleSplit(n_splits=50, test_size=0.2, random_state=0),
"score_type": "both",
}
display = LearningCurveDisplay.from_estimator(model, **params, ...)
La classe affiche automatiquement la courbe d'apprentissage mais vous pouvez appelez la méthode .plot() pour personnaliser votre graphique
Exemple avec un SVC entraîné sur le dataset digits:¶
On charge les données et entraîne un SVC
from sklearn.datasets import load_digits
from sklearn.model_selection import train_test_split
from sklearn.svm import SVC
X, y = load_digits(return_X_y=True)
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.1, random_state=42)
svc = SVC(kernel="rbf", gamma=0.001)
# attention dans la pratique je choisi ici les paramètres déja optimisés
import numpy as np
params = {
"X": X_train,
"y": y_train,
"train_sizes": np.linspace(0.1, 1.0, 5),
"cv": 5,
"score_type": "both",
}
from sklearn.model_selection import LearningCurveDisplay
LearningCurveDisplay.from_estimator(svc, **params);
Utiliser la validation croisée pour évaluer un modèle¶
Rappelons qu'il est toujours souhaitable d'évaluer vos modèles entraînés sur un partie cross validée du data set d'apprentissage X_train (si vous avez assez de données)
Pour obtenir un score moins sensible aux problème du compromis biais/variance, vous pouvez calculer un score cross validé avec la classe cross_validate ou cross_val_score :
cv_results = cross_validate(model, X, y, cv=...)
cv_results['test_score'] # la clé test score_enregistre le score de chaque entrainement cross-validé
Améliorer les données d'entrées¶
La réduction de dimensionalité¶
La sélection de features¶
A venir :)
L'ingénieurie de features¶
A venir : )
Pipelines dans scikit-learn¶
Ce sont des objets permettant d'implémenter une chaîne de traitements modularisés qui facilteront l'automatisation

L'objet pipeline est fabriqué de telle sorte qu'il hérite des méthodes du dernier objet de la séquence:
- pour les Transformers: les méthodes
fitettransform - pour les Modeles: les méhtodes
fit,predict,score, ...
- A l'éxecution du
.fitde la pipeline, la méthodes.fit_transformde chaque transformer est appelée, pour le modèle, seule la méthode.fitest appelée - les outputs des transformers et celle du modèle sont enregistrés dans la mémoire de la pipeline
- A l'éxécution de la méthode prédict, seule les méthodes
.transformdes transformers sont appelées, et la méthode.predictpour le modèle
Cette construction évite de faire du data leakage :)
Avantages
- rend votre workflow plus facilement lisible et compréhensive
- facilite l'automatisation des itérations de votre chaîne de traitement
- rend votre travail plus facilement reproductible et déployable
Exemples d'utilisation¶
Nous utilisons un dataset pour la prédiction d'assurances de santé en fonction de diverses variables
import pandas as pd
import numpy as np
data = pd.read_csv("https://filedn.eu/lefeldrXcsSFgCcgc48eaLY/datasets/regression/data_insurance.csv")
data.head()
X = data.drop(columns='charges')
y = data['charges']
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.33, random_state=42)
Nous allons réaliser les traitements suivants, dans une même pipeline:
- imputation des valeurs manquantes
- scaling des features numériques
- encodage des features catégorielles
- entraintement du modèle
Imputation et scaling¶
from sklearn.pipeline import Pipeline
from sklearn.impute import SimpleImputer
from sklearn.preprocessing import StandardScaler
pipe = Pipeline([
('imputer', SimpleImputer()),
('scaler', StandardScaler())
])
pipe.fit_transform(X_train[['age']])[0:3]
# accéder aux étapes
pipe[0]
pipe['imputer']
Son rôle est d'appliquer des traitements sur des colonnes spécifiques qui vont se faire en parallèle
Dans cet exemple, cela va nous servir à faire l'encodage des features (en one-hot-encoder)
from sklearn.preprocessing import OneHotEncoder
from sklearn.compose import ColumnTransformer
# Imputation et scaling des variables numériques
num_transformer = Pipeline([
('imputer', SimpleImputer()),
('scaler', StandardScaler())])
# Encodage de la feature catégorielle
cat_transformer = OneHotEncoder(handle_unknown='ignore')
# On parallelise les deux traitements "num_transformer" et "cat_transformer"
preprocessor = ColumnTransformer([
('num_tr', num_transformer, ['age','bmi']),
('cat_tr', cat_transformer, ['smoker', 'region'])])
# visualisation des pipelines en HTML
from sklearn import set_config; set_config(display='diagram')
preprocessor
X_train_transformed = preprocessor.fit_transform(X_train)
display(X_train.head(3))
display(pd.DataFrame(X_train_transformed).head(3))
preprocessor.feature_names_in_
# bug dans la version 1.0.2: le transformer SimpleImputer n'a pas encore de méthode get_feature_names_out
SimpleImputer.get_feature_names_out = (lambda self, names=None: self.feature_names_in_)
# Nouveau dans scikit-learn 1.0.2
preprocessor.get_feature_names_out()
pd.DataFrame(X_train_transformed,columns = [preprocessor.get_feature_names_out()]).head()
la variable 'children' n'a pas été traitée par le ColumnTransformer, par défaut elle n'est pas renvoyée
preprocessor
Comme elle n'a pas besoin d'être encodée, on peut la renvoyer telle quelle avec reminder='passthrough'
preprocessor = ColumnTransformer([
('num_tr', num_transformer, ['age','bmi']),
('cat_tr', cat_transformer, ['region','smoker'])],
remainder='passthrough')
preprocessor
pd.DataFrame(preprocessor.fit_transform(X_train)
,columns = [preprocessor.get_feature_names_out()]).head(3)
On peut également appliquer dans une pipeline des fonctions quelconque en les encapsulant grace à FunctionTransformer.
Créons un Transformer pour arrondir les données dans notre dataframe
from sklearn.preprocessing import FunctionTransformer
rounder = FunctionTransformer(lambda array: np.round(array, decimals=2))
rounder.get_feature_names_out = (lambda self, names=None: self.feature_names_in_)
num_transformer = Pipeline([
('imputer', SimpleImputer()),
('scaler', StandardScaler()),
('rounder', rounder)])
preprocessor = ColumnTransformer([
('num_tr', num_transformer, ['bmi', 'age']),
('cat_tr', cat_transformer, ['region', 'smoker'])],
remainder='passthrough')
preprocessor
pd.DataFrame(preprocessor.fit_transform(X_train)).head(3)
Permet d'appliquer des transformers en parallèle et de concatener l'output de chaque transformer à notre data set
Cela peut être utile pour créer de nouvelles features et les ajouter au dataset:
from sklearn.pipeline import FeatureUnion
# On crée une nouvelle variable en en multipliant deux
bmi_age_ratio = FunctionTransformer(lambda df: pd.DataFrame(df["bmi"] / df["age"]))
union = FeatureUnion([
('preprocess', preprocessor), # colonnes 0-8
('bmi_age_ratio', bmi_age_ratio) # nouvelle colonne 9
])
union
Quelques raccourcis¶
from sklearn.pipeline import make_pipeline
from sklearn.pipeline import make_union
from sklearn.compose import make_column_transformer
Pipeline([
('my_name_for_imputer', SimpleImputer()),
('my_name_for_scaler', StandardScaler())
])
# est équivalent à:
make_pipeline(SimpleImputer(), StandardScaler())
num_transformer = make_pipeline(SimpleImputer(), StandardScaler())
cat_transformer = OneHotEncoder()
preproc_basic = make_column_transformer((num_transformer, ['age', 'bmi']),
(cat_transformer, ['smoker', 'region']),
remainder='passthrough')
preproc_full = make_union(preproc_basic, bmi_age_ratio)
preproc_full
On aurait pu aussi utiliser make_column_selector pour sélectionner les colonnes à traiter par leur dtype
X_train.dtypes
from sklearn.compose import make_column_selector
num_col = make_column_selector(dtype_include=['float64'])
cat_col = make_column_selector(dtype_include=['object','bool'])
num_transformer = make_pipeline(SimpleImputer(), StandardScaler())
num_col = make_column_selector(dtype_include=['float64'])
cat_transformer = OneHotEncoder()
cat_col = make_column_selector(dtype_include=['object','bool'])
preproc_basic = make_column_transformer(
(num_transformer, num_col),
(cat_transformer, cat_col),
remainder='passthrough')
preproc_full = make_union(preproc_basic, bmi_age_ratio)
preproc_full
Rajoutons l'entrainement du modèle à notre pipeline¶
Rajout d'un modèle Ridge¶
from sklearn.linear_model import Ridge
# Pipeline de preprocessing
num_transformer = make_pipeline(SimpleImputer(), StandardScaler())
cat_transformer = OneHotEncoder()
preproc = make_column_transformer(
(num_transformer, make_column_selector(dtype_include=['float64'])),
(cat_transformer, make_column_selector(dtype_include=['object','bool'])),
remainder='passthrough')
# Ajout du modèle
pipe = make_pipeline(preproc, Ridge())
pipe
Entrainement et résultats¶
# Preprocessing et entrainement du modèle
pipe.fit(X_train,y_train)
# Prédictions
pipe.predict(X_test.iloc[0:2])
# Score
print(f"Score cross-validé moyen sur le train set: {cross_val_score(pipe, X_train, y_train, cv=5, scoring='r2').mean()}")
print(f"Score sur le test set:{pipe.score(X_test,y_test)}")
Grid Search dans une pipeline¶
On veut vérifier quelle combinaison des paramètres du préprocessing et de l'entrainement donne les meilleurs résultats
On peut pour cela faire un GridSearch sur n'importe quelle composant de la pipeline, avec la syntaxe :
nom_etape__nom_transformer__nom_hyperparam
from sklearn.model_selection import GridSearchCV
# On peut afficher tous les paramètres de tout les composants de la pipeline
pipe.get_params().keys()
pipe.get_params()['columntransformer']
grid_search = GridSearchCV(
pipe,
param_grid={
# grille des hyper paramètres à tester
'columntransformer__pipeline__simpleimputer__strategy': ['mean', 'median'],
'ridge__alpha': [0.1, 0.5, 1, 5, 10]},
cv=5,
scoring="r2")
# entraine toute la pipeline et la ré-entraine avec les meilleurs paramètres trouvés
grid_search.fit(X_train, y_train)
grid_search.best_params_
On enregistre la pipeline entrainée avec les meilleurs estimateurs:
pipe_tuned = grid_search.best_estimator_
Mettre des transformations en cache pour economiser du temps de calcul¶
Certaines opérations d'une pipeline peuvent être mise en cache afin de ne pas être recalculés:
- les calculs des hyperparmètres du modèle
from tempfile import mkdtemp
from shutil import rmtree
# Create a temp folder
cachedir = mkdtemp()
# Instantiate the pipeline with cache parameter
pipe = make_pipeline(preproc, Ridge(), memory=cachedir)
# Clear the cache directory after the cross-validation
rmtree(cachedir)
Débuger sa pipeline¶
# acceder a chacun des composants
pipe_tuned.named_steps.keys()
# vérifier une étape intermédiaire
pipe_tuned.named_steps["columntransformer"].fit_transform(X_train).shape
Etapes finales¶
Exporter sa pipeline entrainée¶
Une fois votre chaine de traitement exécutée, vous avez interêt à sauvegarder l'ensemble de ses traitements. Elle peut être ensuite rechargée, pour faire d'autres iterations, ou la déployer.
Par exemple, je peux sauvegarder ma pipeline complète de traitement et la recharger plus tard pour faire des prédictions
# le module pickle de python permet de sauvegarder n'importe quel objet
import pickle
# spécifier le path pour le fichier final
from pathlib import Path
import os
export_path = Path("/home/nico/code/demos/")
os.path.join(export_path,'test')
# exporter la pipeline
export_path = "/home/nico/code/demos/"
with open(os.path.join(export_path,"pipeline.pkl"), "wb") as file:
pickle.dump(pipe_tuned, file)
# recharger la pipeline
my_pipeline = pickle.load(open(os.path.join(export_path,"pipeline.pkl"),"rb"))
# faire une prédiction avec la pipeline entrainée
my_pipeline.score(X_test, y_test)