Leçon : Evaluation d'un modèle¶
Les métriques d'évaluation¶
Les métriques d'évaluation servent à quantifier la performance d'un modèle (une fois qu'il est entrainé) pour un problème donné, et ce tout au long du cycle de vie d'un modèle.
from sklearn import metrics
Attention !¶
Les métriques sont à choisir en fonction des caractéristiques de la tâche: classification, régression, clustering ...
Une métrique seule ne vous donne qu'un point de vue partiel de la performance d'un modèle pour une tâche : il est souvent souhaitable de comparer différentes métriques d'évaluation
De plus, la métrique ne vous donne pas nécessairement d'information concernant son interprétabilité, il convient d'inspecter son modèle par des analyses complémentaires comme à l'analyse des relations entre la variable de réponse et les features, ou des relations entre les features entre elles.
Aucun modèle ne peut avoir les meilleurs performances pour toutes les tâches (No Free Lunch Theorem) !
Score de référence (baseline)¶
Il convient de toujours de calculer le score d'un modèle de référence pour servir de point de comparaison à votre métrique d'évaluation !
Il peut s'agir d'un résultat provenant de l'état de l'art du domaine étudié, par exemple:
- un modèle physique pour la prédiction du réchauffement climatique
- la performance d'un humain sur la même tache
Ou sinon d'un modèle stupide donnant une réponse stéréotypée, par exemple:
- donner une réponse aléaroire
- pour la classification: prédire la classe la plus fréquente
- pour la régression: prédire une mesure de tendance centrale (moyenne, médiane, mode)
- ...
DummyClassifier pour les classifieurs :
import numpy as np
from sklearn.dummy import DummyClassifier
X = np.array([-1, 1, 1, 1])
y = np.array([0, 1, 1, 1])
dummy_clf = DummyClassifier(strategy="most_frequent")
dummy_clf.fit(X, y)
dummy_clf.predict(X)
dummy_clf.score(X, y)
0.75
DummyClassifier pour les regresseurs :
import numpy as np
from sklearn.dummy import DummyRegressor
X = np.array([1.0, 2.0, 3.0, 4.0])
y = np.array([2.0, 3.0, 5.0, 10.0])
dummy_regr = DummyRegressor(strategy="mean")
dummy_regr.fit(X, y)
dummy_regr.predict(X)
dummy_regr.score(X, y)
0.0
Exemple de data set pour la régression¶
Ce data set mesure la nombre de locations de vélo en fonction de certaines environnementales variables comme la météo :
from sklearn.datasets import fetch_openml
from sklearn.model_selection import train_test_split
bikes = fetch_openml("Bike_Sharing_Demand", version=2, as_frame=True, parser='auto')
bikes.data.head()
| season | year | month | hour | holiday | weekday | workingday | weather | temp | feel_temp | humidity | windspeed | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | spring | 0 | 1 | 0 | False | 6 | False | clear | 9.84 | 14.395 | 0.81 | 0.0 |
| 1 | spring | 0 | 1 | 1 | False | 6 | False | clear | 9.02 | 13.635 | 0.80 | 0.0 |
| 2 | spring | 0 | 1 | 2 | False | 6 | False | clear | 9.02 | 13.635 | 0.80 | 0.0 |
| 3 | spring | 0 | 1 | 3 | False | 6 | False | clear | 9.84 | 14.395 | 0.75 | 0.0 |
| 4 | spring | 0 | 1 | 4 | False | 6 | False | clear | 9.84 | 14.395 | 0.75 | 0.0 |
bikes.target.head()
0 16 1 40 2 32 3 13 4 1 Name: count, dtype: int64
Methode hold out
# Make an explicit copy to avoid "SettingWithCopyWarning" from pandas
X, y = bikes.data.copy(), bikes.target
# Split the data into a training set and a test set
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=0)
Préparation minimale : scaling des features numériques & encoding des features catégorielles
numerical_features = [
"temp",
"feel_temp",
"humidity",
"windspeed",
]
categorical_features = X_train.columns.drop(numerical_features)
from sklearn.compose import ColumnTransformer
from sklearn.preprocessing import OneHotEncoder, QuantileTransformer
mlp_preprocessor = ColumnTransformer(
transformers=[
("num", QuantileTransformer(n_quantiles=100), numerical_features),
("cat", OneHotEncoder(handle_unknown="ignore"), categorical_features),
]
)
mlp_preprocessor
ColumnTransformer(transformers=[('num', QuantileTransformer(n_quantiles=100),
['temp', 'feel_temp', 'humidity',
'windspeed']),
('cat', OneHotEncoder(handle_unknown='ignore'),
Index(['season', 'year', 'month', 'hour', 'holiday', 'weekday', 'workingday',
'weather'],
dtype='object'))])In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
ColumnTransformer(transformers=[('num', QuantileTransformer(n_quantiles=100),
['temp', 'feel_temp', 'humidity',
'windspeed']),
('cat', OneHotEncoder(handle_unknown='ignore'),
Index(['season', 'year', 'month', 'hour', 'holiday', 'weekday', 'workingday',
'weather'],
dtype='object'))])['temp', 'feel_temp', 'humidity', 'windspeed']
QuantileTransformer(n_quantiles=100)
Index(['season', 'year', 'month', 'hour', 'holiday', 'weekday', 'workingday',
'weather'],
dtype='object')OneHotEncoder(handle_unknown='ignore')
Entrainement d'un modèle : MLP Regressor
from sklearn.neural_network import MLPRegressor
from sklearn.pipeline import make_pipeline
print("Training MLPRegressor...")
mlp_model = make_pipeline(
mlp_preprocessor,
MLPRegressor(
hidden_layer_sizes=(30, 15),
learning_rate_init=0.01,
early_stopping=True,
random_state=0,
),
)
mlp_model.fit(X_train, y_train)
Training MLPRegressor...
Pipeline(steps=[('columntransformer',
ColumnTransformer(transformers=[('num',
QuantileTransformer(n_quantiles=100),
['temp', 'feel_temp',
'humidity', 'windspeed']),
('cat',
OneHotEncoder(handle_unknown='ignore'),
Index(['season', 'year', 'month', 'hour', 'holiday', 'weekday', 'workingday',
'weather'],
dtype='object'))])),
('mlpregressor',
MLPRegressor(early_stopping=True, hidden_layer_sizes=(30, 15),
learning_rate_init=0.01, random_state=0))])In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
Pipeline(steps=[('columntransformer',
ColumnTransformer(transformers=[('num',
QuantileTransformer(n_quantiles=100),
['temp', 'feel_temp',
'humidity', 'windspeed']),
('cat',
OneHotEncoder(handle_unknown='ignore'),
Index(['season', 'year', 'month', 'hour', 'holiday', 'weekday', 'workingday',
'weather'],
dtype='object'))])),
('mlpregressor',
MLPRegressor(early_stopping=True, hidden_layer_sizes=(30, 15),
learning_rate_init=0.01, random_state=0))])ColumnTransformer(transformers=[('num', QuantileTransformer(n_quantiles=100),
['temp', 'feel_temp', 'humidity',
'windspeed']),
('cat', OneHotEncoder(handle_unknown='ignore'),
Index(['season', 'year', 'month', 'hour', 'holiday', 'weekday', 'workingday',
'weather'],
dtype='object'))])['temp', 'feel_temp', 'humidity', 'windspeed']
QuantileTransformer(n_quantiles=100)
Index(['season', 'year', 'month', 'hour', 'holiday', 'weekday', 'workingday',
'weather'],
dtype='object')OneHotEncoder(handle_unknown='ignore')
MLPRegressor(early_stopping=True, hidden_layer_sizes=(30, 15),
learning_rate_init=0.01, random_state=0)Mean Squared Error (MSE)¶
Elle mesure la différence quadratique moyenne entre les labels $y$ et leurs valeur prédites $\hat y$ :
Quelques une de ses caractéristiques:
- utile pour pénaliser les erreurs importantes
- très sensible aux outliers
- ne donne pas la direction de l'erreur
- ne donne pas le même ordre de grandeur que $y$
Root Mean Squared Error (MSE)¶
L'utilisation de la racine carrée permet d'avoir une erreur de même ordre de grandeur que les labels
Mean Absolute Error (MAE)¶
Elle mesure la moyenne de la différence absolue (norme $L_1$) entre les labels $y$ et leurs valeur prédites $\hat y$ :
Elle est moins sensible aux outliers, utilisez la si les erreurs (petites ou grandes) ont la même importance
Max Error¶
Elle mesure la plus grande erreur faite par le modèle:
Utilisez Max error lorsque vous souhaitez limiter la magnitude des erreurs
Le coefficient de détermination $R^2$¶
Mesure la proportion de variance observée sur $y$ qui est expliquée par les variables $X$ du dataset. Elle évalue la qualité de l'ajustement du modèle (goodness of fit) par rapport à un modèle stupide qui prédirait toujours $\bar y$
- $R^2$ = 1 caratérise modèle qui ajuste parfaitement les données
- $R^2$ ~ 0 caractéirse un modèle qui ne fait pas mieux que le modèle stupide
- $R^2$ < 0 caractérise un modèle plus mauvais encore !
Quelques une de ses caractéristiques:
- donne une mesure d'erreur standardisée (entre -1 et 1)
- peux être utilisée pour comparer la performance d'un modèle sur différents data set
Exemples de comparaison de métrique durant la cross-validation¶
from sklearn.model_selection import cross_validate
cv_results = cross_validate(mlp_model, X_train, y_train, cv=5,
scoring = ['neg_mean_absolute_error',
'neg_mean_squared_error',
'max_error','r2'])
pd.DataFrame(cv_results)
| fit_time | score_time | test_neg_mean_absolute_error | test_neg_mean_squared_error | test_max_error | test_r2 | |
|---|---|---|---|---|---|---|
| 0 | 1.698149 | 0.010826 | -30.098396 | -2280.340766 | -360.056151 | 0.929707 |
| 1 | 5.293706 | 0.020174 | -28.083844 | -1998.342875 | -463.954552 | 0.939888 |
| 2 | 4.315770 | 0.013060 | -27.319143 | -1836.767237 | -271.759472 | 0.942871 |
| 3 | 3.086931 | 0.009863 | -28.370111 | -2141.842609 | -443.785465 | 0.936017 |
| 4 | 3.279214 | 0.011184 | -27.561188 | -1887.310100 | -364.704323 | 0.942186 |
cv_results['test_r2'].mean()
0.938133980593818
On distingue souvent 3 types de classification: binaire {$C_0$,$C_1$}, multiclasse {$C_1 \ldots C_n $}, et mutlilabel { {$C_1$,$D_1$},$\ldots$,$C_n$,$D_n$} }
Matrice de confusion¶
Dans les tâches de classification, on distingue souvent deux types d'erreurs, que l'on peut représenter par une matrice de confusion
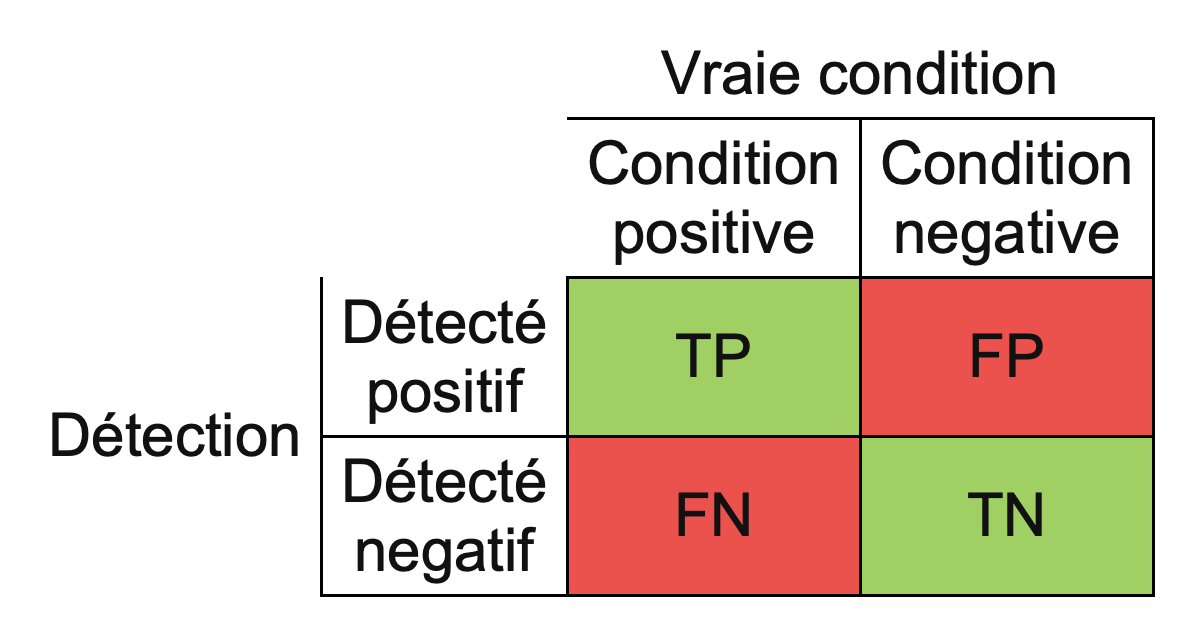
Exemple de matrice de confusion pour une classification multiclasse¶
on entraine un SVC sur le très connu iris data set:
import matplotlib.pyplot as plt
from sklearn import svm, datasets
from sklearn.model_selection import train_test_split
from sklearn.metrics import ConfusionMatrixDisplay
# import some data to play with
iris = datasets.load_iris()
X = iris.data
y = iris.target
class_names = iris.target_names
# Split the data into a training set and a test set
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=0)
# Run classifier, using a model that is too regularized (C too low) to see
# the impact on the results
classifier = svm.SVC(kernel="linear", C=0.01).fit(X_train, y_train)
On représente souvent la matrice de confusion sous forme de carte de chaleur:
confmat = ConfusionMatrixDisplay.from_estimator(
classifier,
X_test,
y_test,
display_labels=class_names,
cmap=plt.cm.Blues,
)
confmat.ax_.set_title("Confusion matrix");
Beaucoup de métriques de classification se basent sur la matrice de confusion ! (voir la page wikipédia dédiée pour plus de détails)
Accuracy score¶
C'est le score le plus simple, il correspond à la fraction de réponses correcte:
Attention ! C'est une mesure qui donne un score trop confiant en particulier lorsque le dataset à traiter contient des classes déséquilibrées, on lui préfera le balanced accuracy score dans ce cas
Balanced accuracy score¶
Recall / sensitivity / true positive rate¶
Cette métrique mesure la capacité du classifieur à détecter les vrais positifs parmi les échantillons positifs:
On utilise de préférence le recall lorsqu'il est important d'identifier les occurences d'une classe, par exemple pour un test de dépistage d'une maladie
Precision¶
Cette métrique mesure la capacité du classifieur à détecter les vrais positifs parmi les prédictions positives:
On utilise de préférence la précision lorsqu'il est important d'identifier correctement une classe, par exemple lorsque l'on utilise un moteur de recherche
F-score¶
Il s'agit d'une métrique combinant, avec un poids, la précision et le recall. On utilise souvent le F1 score :
mais on peut généraliser la pondération avec n'importe quelle valeur:
- le meilleur score de $F_\beta$ vaut 1, le pire score 0
- donne le même poids au recall et la précision, si $\beta > 1$ on favorise le recall
Cette fonction de scikit-learn renvoie un rapport intégrant plusieurs métriques de classification:
- la précision
- le recall
- le F1-score
- le support (le nombre de points utilisés)
from sklearn.metrics import classification_report
print(classification_report(y_test,classifier.predict(X_test) , target_names=class_names))
precision recall f1-score support
setosa 1.00 1.00 1.00 13
versicolor 1.00 0.62 0.77 16
virginica 0.60 1.00 0.75 9
accuracy 0.84 38
macro avg 0.87 0.88 0.84 38
weighted avg 0.91 0.84 0.84 38
Specificity / selectivity / true negative rate¶
Cette métrique mesure la capacité du classifieur à détecter les vrais négatifs parmi les échantillons négatifs:
La courbe ROC (Receiver Operating Characteristic) est une mesure de la performance d'un classifieur binaire, qui provient de la théorie de la détection du signal (elle etait utilisée pour séparer les signaux radar du bruit de fond)
Pratiquement, elle est calculée en faisant calculant le True positive rate (ou sensitivity) versus le True negative rate (1-specificity) en faisant varier un seuil de discrimination. La métrique consiste alors à mesurer l'aire sous la courve ROC (AUC) comme score

Pour en avoir un exemple interactif pour comprendre sa construction, consulter ce site
Bien que certaines des métriques présentées peuvent avoir des variantes multilabel, il existe des métriques spécifiques aux tâches de classification multilabel (qui consistent à prédire plusieurs labels en même temps)
Certaines d'entre elles sont décrites et implémentées dans scikit-learn comme la coverage error, label ranking average precision, la ranking loss, ...
Métriques de clustering¶
Métriques nécessitant la connaissance de labels¶
La plupart des métriques d'évalutation du clustering nécessitent la connaissance à priori de labels
Comment se fait il, me direz vous ? l'apprentissage non supervisé n'est-il pas sensé se faire sans la connaissance de la target y ? 🤔
Dans la pratique, de nombreux algorithmes de clustering peuvent assigner aux points du dataset un label (le numéro du cluster auquel ils appartiennent). On peut alors utiliser ce label pour calculer la métrique d'évaluation
Connaissant l'assignement entre les labels labels_pred assignés à chaque point de donnée par le modèle de clustering et leur label réel label_true, ce type de métrique mesure la similarité entre deux assignements (mesurée entre 0 et 1), en ignorant les permutations
labels_true = [0, 0, 0, 1, 1, 1]
labels_pred = [0, 0, 1, 1, 2, 2]
metrics.rand_score(labels_true, labels_pred)
0.6666666666666666
le score ne change pas si on permute uniquement les valeurs des labels:
labels_pred = [1, 1, 0, 0, 3, 3]
metrics.rand_score(labels_true, labels_pred)
0.6666666666666666
Cependant le Rand index n'assure pas d'obtenir une valeur nulle pour une assignation dûe au hasard. l'adjusted Rand Index corrige ce problème en prenant le niveau de chance dû au hasard comme référence
metrics.adjusted_rand_score(labels_true, labels_pred)
0.24242424242424243
Les métrique de type Information Mutuelle¶
Ce type de métriques provient de la théorie de l'information. Elle mesure l'information mutuelle, c.a.d l'agrément entre les labels labels_pred assignés à chaque point de donnée par le modèle de clustering et leur label réel label_true
Par exemple, l'Adjusted MutualInformation qui prend en compte le hasard, renvoie un score compris entre 0 (pour deux partition assignée dont on aurait assigné les labels au hasard) et 1 pour deux partitions parfaitement identiques
labels_true = [0, 0, 0, 1, 1, 1]
labels_pred = [0, 0, 1, 1, 2, 2]
metrics.adjusted_mutual_info_score(labels_true, labels_pred)
0.2987924581708901
Quelques métriques agnostique des labels¶
Contrairement aux métriques présentées plus tôt, certaines métriques de clustering ne nécessitent pas la connaissance d'un label label_true.
Intuitivement elles vont s'appuyer sur une évaluation de l'homogeneité des clusters produits
Le score de silhouette est une métrique basée sur la distance entre un points et tout ceux d'un même cluster, en comparaison avec tout ceux du cluster le plus proche.
Il se base sur deux scores :
- a : la distance moyenne entre un point et les autres points du même cluster
- b: la distance moyenne entre un point et les autres points du cluster voisin le plus proche
Il varie entre -1, indiquant un clusterting fortement incorrect et 1 pour des clusters très dense et distincts les uns des autres,
Exemple avec un K-means appliqué au data set iris:
from sklearn import datasets
X, y = datasets.load_iris(return_X_y=True)
from sklearn.cluster import KMeans
kmeans_model = KMeans(n_clusters=3, random_state=42, n_init='auto').fit(X_train)
labels = kmeans_model.labels_
metrics.silhouette_score(X_train, labels, metric='euclidean')
0.5618512101716054
Il s'agit d'une métrique basée sur le ratio entre; la somme de la dispersion intra-clusters et celle inter-clusters
Cette métrique n'est pas normalisée et donne en général un nombre réel positif, d'autant plus grand que les clusters sont disjoints et homogènes :
metrics.calinski_harabasz_score(X_train, labels)
433.0933726899339
Il s'agit d'une métrique basée sur la similarité moyenne entre les clusters (mesurée par le ratio de la distance intra-clusters à celle inter-clusters)
Le score minimum est zéro, un score faible indiquant un clustering de meilleure qualité
from sklearn.metrics import davies_bouldin_score
davies_bouldin_score(X_train, labels)
0.6402727236753957
Analyse des relations entre variables¶
Utiliser une ou plusieurs métriques est un premier pas, mais ne suffit pas à comprendre les relations entre les variables et leurs impacts sur les prédictions.
Analyse des relations entre features¶
Il s'agit d'analyser les relations entre les features, par exemple en examinant l'importance des features utilisée par le modèle.
Certains modèles possèdent nativement un tel score :
- les coefficients d'une régression
- l'importance des features utilisées dans les partition d'un arbre de décision
La plupart des modèles n'ayant pas de score natifs, on peut utiliser des méthodes génériques:
- l'importance des features calculées par permutation (en particulier pour les modèles non linéaires sur des données tabulaires)
- des modules dédiés proposent des methodes de calcul d'importance des features, commeLIME ou SHAP
Analyse des relations entre features et variable cible¶
Il s'agit d'analyser les relations entre les prédicteurs et variables cibles, en particulier leur degré de dépendance
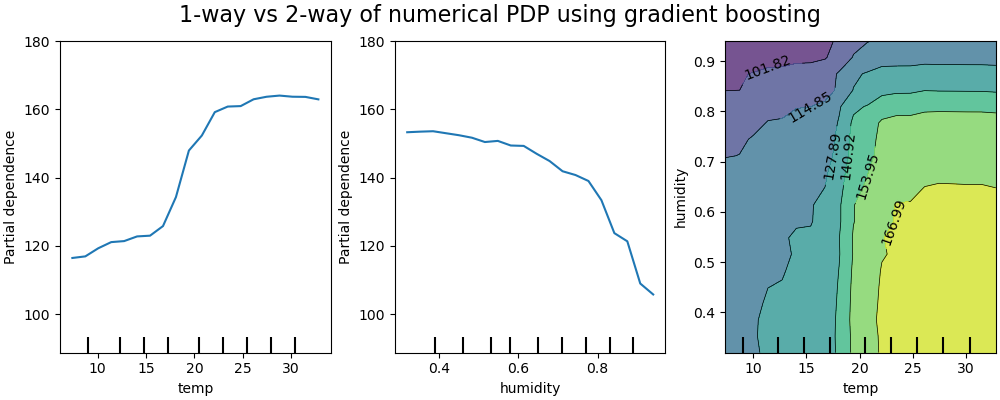
Analyse de dépendance partielle (partial dependence plots - PDP)¶
On utilise souvent les graphiques PDP pour représenter la dépendance de la variable de réponse, avec un sous groupe de features d'intérêt (généralement un petit nombre parmi les plus importantes), en comparaison avec toutes les autres features
Exemple de PDP sur un modèle de prédiction de la location de vélo avec les variables température et humidité
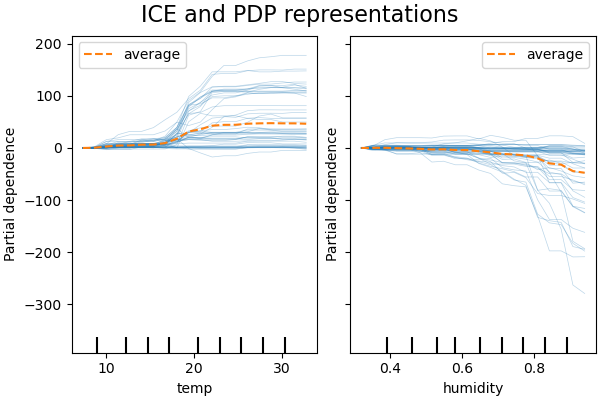
l'ICE représente la dépendance entre la variable de réponse et un sous groupe de features représentatif, pour chaque obervation séparément. On représente généralement une feature d'intrêt par grapique d'ICE (pour faciliter sa lecture)
Exemple d'ICE sur le même modèle de prédiction de location de vélo:
Pour plus de détails sur l'analyse de dépendence partielle des variables de ce data set, vous pouvez voir l'exemple complet sur scikit-learn